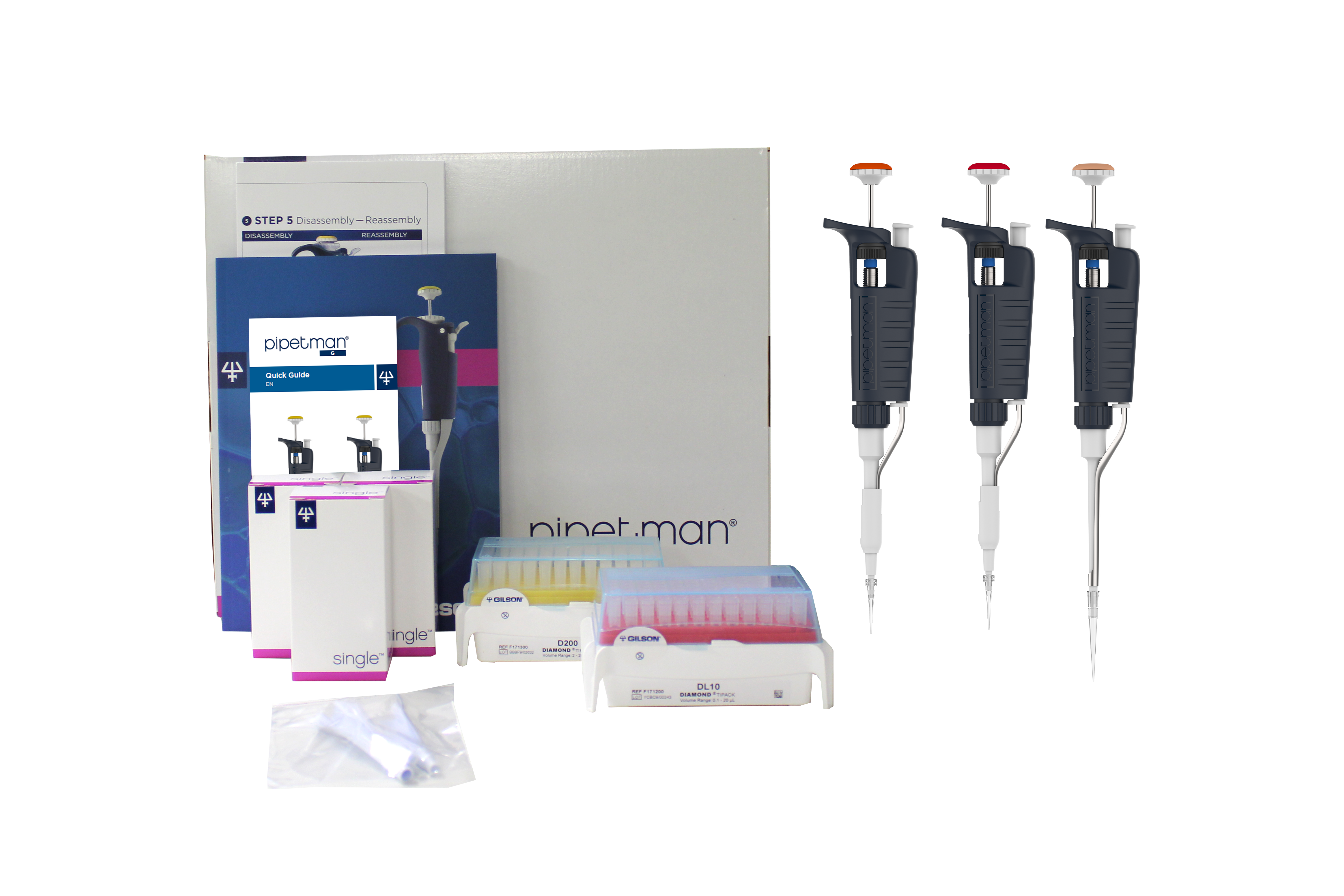
製品情報
修理・サービス
修理/バリデーション/校正サービスはもとより、リキッドハンドリング講習会、会員制サイト 「ピペットマンクラブ」など、ピペットマンを末永く安心してご使用いただくための各種サービス・サポートメニューをご提供しています。
製品を末永くご愛用頂くための日常メンテナンス、キャリブレーションおよび管理に関する3種類のコースによる講習会を開催しております。
専門スタッフが迅速・正確なアフターサービスをおこなっております。 弊社キャリブレーションルームは ISO 8655 規格準拠の測定環境と設備を完備しています。